3.1 ORGANISMOS PROCARIÓTICOS
Los seres vivos integrados por células procariotas
constituyen actualmente el reino de los monerados (o también protistas
inferiores). El resto de los seres
vivos, ya sean unicelulares (protistas) o pluricelulares (vegetales o metafitas
y animales o metazuarios), están constituidos por una o muchas células
eucariotas o eucitos, tienen núcleo normal
En general, los genomas bacterianos tienen un tamaño
inferior a 5 Mb, aunque en el caso de Bacillus megaterium el genoma tiene un
tamaño de 30 Mb. La mayor parte del genoma corresponde a genes.
3.1.1 ADN CIRCULAR
GENOMAS PROCARIÓTICOS
Genomas procarióticos
El genoma de la mayoría de los procariontes está formado por
un único cromosoma. Normalmente es una molécula de DNA de doble cadena cerrada
y circular. Existen excepciones como la bacteria Borrelia burgdorfei, cuyo
cromosoma es una cadena lineal!. Algunos genomas bacterianos están formados por
varios cromosomas distintos.
Los genes bacterianos estan muy agrupados, con distancias intergénicas
muy pequeñas, y los intrones son muy escasos.
3.1.2 PROTEÍNAS ASOCIADAS
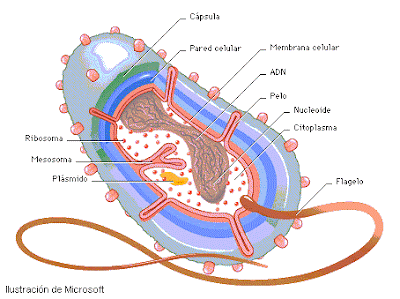
El DNA en bacterias se
organiza en un nucleoide, que se forma por un superenrrollamiento.
El DNA en bacterias se
organiza en un nucleoide, que se forma por un supernrrollamiento.
Superenrollamiento
(supercoiling)
. Superenrollamiento puede ser positivo (p.e. arqueas) o
negativo (eubacterias).
- Superenrollamiento (-): la molécula torsionada gira hacia
la derecha
- Superenrollamiento (+): gira a la izquierda.
El superenrollamiento es una forma de liberar la tensión
torsional producida por la adición (+) o sustracción (-) de vueltas en una
molécula circular de DNA.
El superenrollamiento en E. coli está regulado por unos
enzimas denominados DNA girasa y topoisomerasa I.
El nucleoide es un cromosoma circular superenrrollado.
En E.coli existen una serie de proteinas relacionadas con el
empaquetamiento del DNA que reciben el nombre de proteinas similares a histonas
p.e. histone-like nucleoid structuring protein (H-NS), integration host factor
(IHF) etc.
su similitud con las histonas de los eucariotas es muy baja
(a excepción de las proteinas HU).
Estas proteinas posiblemente son capaces de remodelar el
grado de compactación del DNA del nucleoide, influyendo sobre la expresión
génica.
3.1.3 ADN EXTRACROMOSÓMICO.
3.1.3.1 PLÁSMIDOS.
Genomas plásmidicos
Las células bacterianas contienen comúnmente pequeños
elementos de DNA que no son esenciales para las operaciones básicas de la
célula. Estos componentes se denominan plasmidos.
Los plasmidos no pueden vivir fuera de la célula.
Los plasmidos bacterianos contienen a menudo genes que son
útiles para la célula huésped, p.e. confieren resistencia a antibióticos, promueven la fusión o producen
toxinas.
los plasmidos bacterianos suelen ser circulares, aunque se
han encontrado algunos lineales.
Existencia autónoma del cromosoma bacteriano (raramente se
insertan).
Portadores de genes no presentes en el cromosoma bacteriano
p.e. genes de resistencia a antibióticos.
Un mismo plásmido puede hallarse en especies distintas de
bacterias.
3.1.3.2 BACTERIÓFAGOS
Los bacteriófagos (fagos) son parásitos intracelulares
obligados que se multiplican al interior de las bacterias, haciendo uso de
algunas o todas sus maquinarias biosintéticas (los virus que infectan
bacterias).
Los fagos pueden generar el ciclo lítico o el ciclo
lisogénico, aunque muy pocos son capaces de llevar a cabo ambos.
En el ciclo lítico, las células hospedadoras del fago son
lisadas (destruidas) tras la replicación y encapsulación de las partículas
virales, de forma que los nuevos virus quedan libres para llevar a cabo una
nueva infección.
Por el contrario, en el ciclo lisogénico no se produce la
lisis inmediata de la célula. El genoma del fago puede integrase en el ADN
cromosómico de la bacteria hospedadora, replicándose a la vez que lo hace la
bacteria o bien puede mantenerse estable en forma de plásmido, replicándose de
forma independiente a la replicación bacteriana.
En cualquier caso, el genoma del fago se transmitirá a toda
la progenie de la bacteria originalmente infectada. El fago queda así en estado
de latencia hasta que las condiciones del medio se vean deterioradas:
disminución de nutrientes, aumento de agentes mutagénicos, etc. En este
momento, los fagos endógenos o profagos se activan y dan lugar al ciclo lítico
que termina con la lisis celular.
3.1.3.3 TRANSPOSONES
Los transposones son fragmentos de DNA que pueden pasar de
un cromosoma a otro sin una etapa de existencia independiente. Fueron
descubiertos por Barbara Mc Clintock en
la década de los 40-50, en el genoma del maíz. Son elementos del genoma del
maíz que tenían la capacidad de movilizarse favoreciendo la aparición de
determinados fenotipos
La transposición es el fenómeno por el cual un elemento de
DNA cambia de ubicación física en el cromosoma sin aprovechar ningún tipo de
homología de secuencia, sino a través de una proteína específica denominada
transposasa. Las características que distinguen la transposición de la
recombinación son:
No requiere homología de DNA entre la secuencia dadora y la
aceptora, aunque hay puntos calientes en regiones ricas en AT
Se produce una duplicación de la secuencia aceptora (entre 3
y 12 pb) antes y detrás del transposón
la intervención de la transposasa y, en algunos casos, la
resolvasa.
La transposición puede reestructurar drásticamente la
organización del cromosoma hospedador. Además, puede alterar la expresión de un
gen, bien inactivándolo, bien sobreexpresándolo.
Su presencia la demostró Barbara McClintok en los años 50.
Transposones procarióticos
- Tipos
La clasificación se basa principalmente en los genes que
aparecen
Tipo I
Se conocen como transposones simples o secuencias de
inserción (IS) lo que en 700 o 2000 pb contienen el gen TnpA que codifica la
transposasa flanqueado por dos secuencias invertidas repetidas cortas (15 a 25
pb).
Tipo II
Contienen al menos tres genes: una transposasa (TnpA), una
resolvasa (TnpR) y un gen que suele ser de resistencia a antibióticos. Eso se
encuentra flanqueado por secuencias repetidas invertidas (IR) a la izquierda
(IR-L) y a la derecha (IR-R).
Tipo III
Aquellos fagos que, en lugar de insertarse en el genoma por
recombinación —lo normal—, lo hace mediante transposición. El más conocido es
el fago μ.
La gran mayoría de los transposones eucarióticos utilizan
RNA como intermediario de la transcripción.
3.2 ORGANISMOS EUCARIÓTICOS
Son aquellos en cuyas células puede diferenciarse un núcleo
que contiene el material genético separado de un citoplasma en el que se
encuentran diferentes orgánulos celulares.
Los organismos eucariotas incluyen algas, protozoos, hongos,
plantas superiores, y animales. Este grupo de organismos posee un aparato
mitótico, que son estructuras celulares que participan de un tipo de división
nuclear denominada mitosis; tal como imnúmeras organelas responsables de
funciones específicas, incluyendo mitocondrias, retículo endoplasmático y
cloroplastos.
Las especies eucarióticas se clasifican como diploides (dos
series de cromosomas en el núcleo), o son haploides (1 sola serie de cromosomas
en el nucleo), la mayoría de algas y hongos son haploides y el resto de
eucariotas (incluyendo plantas y animales) son diploides.
La letra n se utiliza para designar el número de cromosomas
de un genoma nuclear de un organismo, la condición diploide es 2n y la haploide
n.
Las condiciones 3n, 4n, 5n, etc se conocen como poliploides.
En una célula diploide, puesto que hay dos series
cromosomitas, existen dos cromosomas de cada tipo. Los miembros de cada par se
conocen como cromosomas homologos. Los miembros de un par homologo básicamente
son identicos y aportan los mismos genes, aunque muchas veces diferentes alelos
(formas diferentes de un mismo gen).
Para determinar el número de cromosomas de una célula, estos
simplemente se tiñen y se cuentan al microscopio óptico.
En los organismos eucarióticos, la mayoría de los genes se
encuentra en los cromosomas del núcleo.
3.2.1 ADN LINEAL Y EMPAQUETAMIENTO
Cada molécula de ADN se empaqueta en un cromosoma el total
de la información genética contenida en los cromosomas de un organismo,
constituye su genoma.
Los cromosomas de una célula, en mitosis o fase M, se
encuentran muy condensados y son transcripcionalmente activos.
En la interfase, están mucho menos condensados y son activos
dirigiendo continuamente la síntesis de RNA.
Cada molécula de ADN que forma un cromosoma ha de contener
un centromero, dos telómeros y varios orígenes de replicación.
Para que una molécula de ADN forma un cromisoma funcional ha
de ser capaz de dirigir la sintesis de ARN y de propagarse, transmitiendose
eficasmente de una generación a otra.
Necesitan un origen de replicación, centrómero que une la
molécula de ADN que lo contiene al huso mitótico durante la divición celular
telómero eciste uno de ellos encada extremo del cromosoma lineal.
Una célula humana contiene alrededor de 2 metros de DNA (1
metro por cada serie cromosomica). El cuerpo humano está constituido por unas
1013 células y cada célula es diploide, por lo que contiene en total unos
2x1013 metros de DNA.
3.2.1.1 HISTONAS
Las proteínas celulares más frecuentes son las proteínas
histonas, siendo que cada célula eucariótica presenta varios cientos de
millones de moléculas de histonas, mientras que las demás proteínas no alcanzan
unos cientos (como mucho, a miles). Son proteínas de masa molecular baja,
aproximadamente 11-12 Kd y exhiben un alto contenido, cerca de 20%, de lisina y
arginina (aminoácidos básicos). Con las cargas positivas de las cadenas
laterales de estos restos, las histonas (que son extremadamente básicas) se
unen a los grupos fosfato del ADN (cargados negativamente); para ello no es
relevante la secuencia de bases dentro del ADN. A menudo, las histonas son
modificadas por metilaciones, acetilaciones, fosforilaciones o
ADP-ribosilaciones.
En los seres humanos hay cinco tipos principales: la histona
H1 y las histonas H2A, H2B, H3 y H4. Estas últimas se denominan también
histonas nucleosomales y forman un octámero con dos histonas de cada; alrededor
de este núcleo se enrolla dos veces un hilo de ADN. Este complejo ADN-histona
recibe el nombre de nucleosoma y constituye el componente primario del
cromosoma.
El empaquetamiento ocurre en el núcleo donde el DNA se
condensa en 46 cromosomas.
La mezcla completa de materiales de los que se compone el cromosoma se conoce
como cromatina. Se trata de DNA y proteínas.
Las histonas son proteínas asociadas al DNA en los
nucleososmas. Los nucleosomas (10 nm) están formados por un octamero compuesto
por dos unidades de cada una de las histonas (H2A, H2B, H3 Y H4).
3.2.1.2 SOLENOIDES
El DNA (asociado a las histonas) da dos vueltas alrededor de
cada octamero de los nucleosomas, el nucleosoma es una forma distendida, de una
forma muy enrollada denominado solenoide (30 nm) , el solenoide mantiene su
forma mediante otra histona H1.
Para conseguir el primer nivel de empaquetamiento el DNA se
enrolla alrededor de las histonas, que actúan, como bobinas de un hilo, un
nuevo enrollamiento genera la conformación del solenoides.
3.2.1.3 CROMOSOMAS
Enrollamientos de orden superior.
Los cromosomas se encuentran muy enrollados, si un solenoide
tiene de diámetro 30 nm, un cromosoma condensado tiene 700 nm. Para esto el
solenoide se enrolla sobre un esqueleto proteico compuesto de la enzima
topoisomerasa II, que es capaz de pasar una cadena de DNA a través de otra.
COMO SE EMPAQUETA EL ADN.
1) el DNA se enrolla sobre los nucleosomas que actúan como
bobinas de hilo.
2) los nucleosomas se enrollan formando un solenoide.
3) el solenoide forma lazos anclados a un esqueleto central.
4) el esqueleto unido a los lazos se dispone como un
solenoide gigante.
3.2.2 COMPLEJIDAD DEL GENOMA
Las principales regiones de un gen
un gen es una región de DNA cromosómico que puede
trascribirse en una molécula de RNA funcional en el momento y lugar adecuados
del proceso de desarrollo de un organismo.
El hecho de definir con precisión que es un gen puede
dificultarse ya que muchos genes eucarioticos contienen segmentos de DNA,
llamados intrones, que se encuentran intercalados en la región transcrita del
gen. Los intrones no contienen información para al formación del producto
génico correspondiente. (p.e proteína). Se transcriben junto con las regiones
codificantes (llamadas exones) pero luego son eliminados del transcrito
inicial.
La correcta secuencia de nucleótidos de los intrones, de la
región reguladora y de la región codificante es necesaria para crear un
trascrito del tamaño adecuado, en el momento y lugar adecuado y estas tres
partes deben considerarse como una unidad funcional completa, en otras palabras
como parte de un gen.
Los análisis de secuencia han demostrado que hay DNA entre
los genes, de función desconocida la mayor parte. El tamaño y la naturaleza de
este DNA dependen del genoma. En hongos, este DNA intergénico es pequeño, pero
en mamíferos es muy grande.
Los tamaños de los genomas se miden en unidades formadas por
miles de bases de nucleotidos (llamados kilobase, kb) o millones de pares de
nucleotidos (megabases, mb),
3.2.3 ADN MITOCONDRIAL.
Los cromosomas de mitocondrias y cloroplastos son de DNA de
doble cadena.
Las mitocondrias se
encuentran en todos los seres eucariotas aerobios; contienen las enzimas para
la mayor parte de las reacciones oxidativas que generan energia para las
funciones celulares.
Actualmente se conocen las secuencias completas del ADN de
varios genomas mitocondriales. Al ADN mitocondrial se lo conoce como ADNmt
(mtDNA).
La estructura del genoma mitocondrial es circular como es el
del genoma bacteriano. Se trata de una mol. circular de ADN, helicoidal, con
doble hebra, y supercondensada. Se conocen algunos pocos casos de genomas
mitocondriales de forma lineal. En muchos casos, el contenido de GC
(guanina-citosina) del ADNmt difiere en gran medida del ADN nuclear, y esto
permite separar el ADNmt del nuclear por centrifugacion en un gradiente de cloruro de cesio.
En los animales, el genoma mitocondrial generalmente es
menor a 20 kb (kilobases); por ej. en el hombre el ADNmt tiene 16.569 bp (pares
de bases). Por su parte, el ADNmt de una levadura contiene cerca de 80.000 bp
(80 kb), mientras que en las plantas varia entre 100 y 2.000 kb.
3.3 ORGANIZACIÓN GENÓMICA VIRAL.
Los genomas virales son muy distintos entre si, muchos estan
compuestos de ADN, que cuando estan empaquetados puede ser de cadena sencilla y
cadena doble. Algunos virus como el HIV (retrovirus) contiene genomas de RNA,
algunos de cadena sencila y otros de cadena doble. Algunos genomas virales
contienen DNA y RNA circulares.
Independiente del genoma del virus hay siempre una fase
intracelular del ciclo infectivo, en la cual este genoma se convierte en DNA de
cadena doble.
Los virus pueden tener DNA duplex, DNA monocatenario, RNA
monocatenario o RNA duplex.
El tamaño de los virus está comprendido entre 20 y 300 nm.
Ya que la mayoría miden menos de 250 nm, límite de resolución del microscopio
óptico, sólo son visibles con ayuda del microscopio electrónico.
Los virus están compuestos de un núcleo central formado por
ácido nucleico (DNA o RNA, pero nunca los dos en el mismo virión) rodeado por
una proteína que constituye la cápsida. El núcleo central y la cápsida forman
conjuntamente la nucleocápsida del virión.










.JPG)

.jpg)









.jpg)
